Stanford大の教材CS231nを使ってNNやCNNを学んでいる.
本記事では,Neural Network(NN)を中心に扱う.
Setting up the data and the model
- Linear Classification用からNN用へscore functionの拡張
- linear mappingを繰り返しの中にnon-linearityを織り交ぜる
Data Preprocessing
- 入力データ
に対して3つの代表的な前処理がある
- Mean subtraction
- 個々の特徴から平均値を引く処理
- 幾何的に解釈すると、データの集まりの中心を原点に持ってくる処理でもある
- Nomalization
- 次元を正規化すること
- 2つの代表的な方法がある
- 標準偏差で入力データを割る方法
- 最小値と最大値を-1~1にする方法
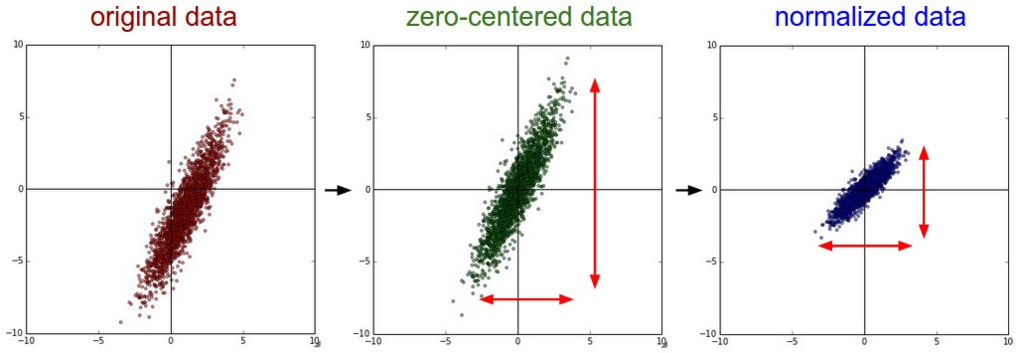 (CS231nより引用)
(CS231nより引用)
- PCAとWhitening
- (前提としてmean subtractionされている状態とする)
- 共分散行列を計算する
# Assume input data matrix X of size [N x D] X -= np.mean(X, axis = 0) # zero-center the data (important) cov = np.dot(X.T, X) / X.shape[0] # get the data covariance matrix
- PCAとWhitening(続き)
- 共分散行列
- (i,j)要素はi行目とj列目のデータの共分散を保持
- 求めた共分散行列から特異値分解する
- 共分散行列
U,S,V = np.linalg.svd(cov)
- PCAとWhitening(続き)
- 入力データをdecorrelateする
Xrot = np.dot(X, U) # decorrelate the data
- PCAとWhitening(続き)
Xrot_reduced = np.dot(X, U[:,:100]) # Xrot_reduced becomes [N x 100]
- whitening
- 全次元に対して固有値で割ってスケールを正規化すること
- もともとmultivariable gaussianだったデータの広がりを、0平均、identitity covariance matrixをもつgaussianとする操作でもある
# whiten the data: # divide by the eigenvalues (which are square roots of the singular values) Xwhite = Xrot / np.sqrt(S + 1e-5)
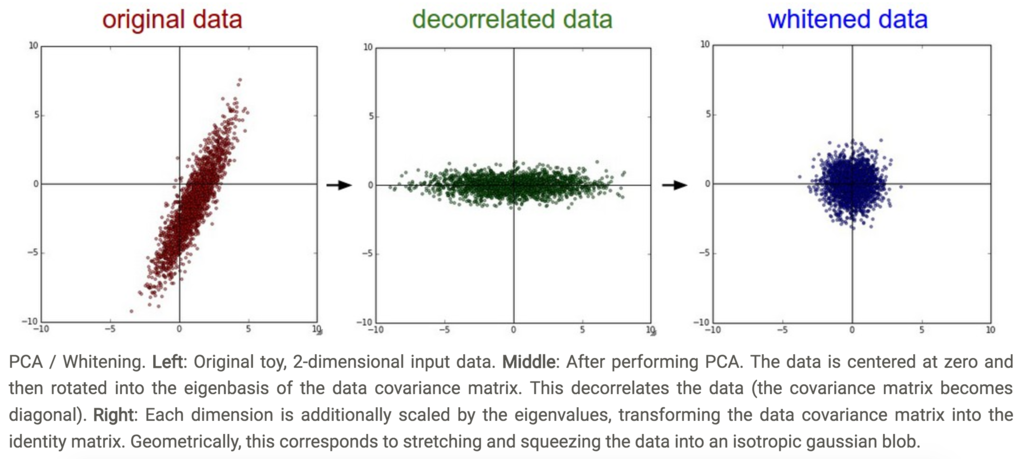 (CS231nより引用)
(CS231nより引用)
- この正規化処理によるデータの変化をCIFAR-10で検証したのが下図
 (CS231nより引用)
(CS231nより引用)
- 落とし穴
- 前処理の統計処理はtraining dataのみで計算して、その後でvalidation dataやtest dataに適用すること
- 全体に対して計算処理して、あとからtraining/validaiton/testと分割してはいけない
Weight Initialization
- 重みを0で初期化してはいけない
- 全neuronが同じ出力をしてしまい、同じ勾配を逆伝播時に算出し、同じようにパラメータを更新してしまう
- 小さい値で初期化する
- なぜ??
- 分散を1/sqrt(n)により補正する
- neuronの出力の分散を1にするため、重みに1/sqrt(n)掛ける
- sparse initialization
- initializing the biases
- バイアスは0に初期化してよい
asymmetry breakingは重みの方で行えばよい
- バイアスは0に初期化してよい
- 実用上の方策としては、ReLUユニットを使い、
pythonw = np.random.randn(n) * sqrt(2.0/n)(He et al.)とすればよい
- Batch Normalization
- 全結合層の後にBatchNorm層を置く
- 初期化に失敗したNNでも、いい結果を残せるかもしれない
Regularization
- NNの過学習を防ぐ方法は幾つかある
- L2 regularization
- L1 regularization
- max norm constraints
- dropout
- L2 regularization(L2正規化)
- NNの各重み
に
を掛ける
は正規化の強さ
- NNの各重み
- L1 regularization(L1正規化)
- 各重み
に
を足す
- 各重み
- L1とL2を組み合わせることも可能(Elastic net regularization)
- L1はzeroに向かわせるはたらきをする
- L2はノイズを混ぜるはたらきをする
- max norm constraints
- 重みの最大値を制限してしまう方法
- 各ニューロンの重みベクトル
に対して、
を満たすようにする
- dropout
- neuronの中の一部を選択的にdeactivateする(下図参照)
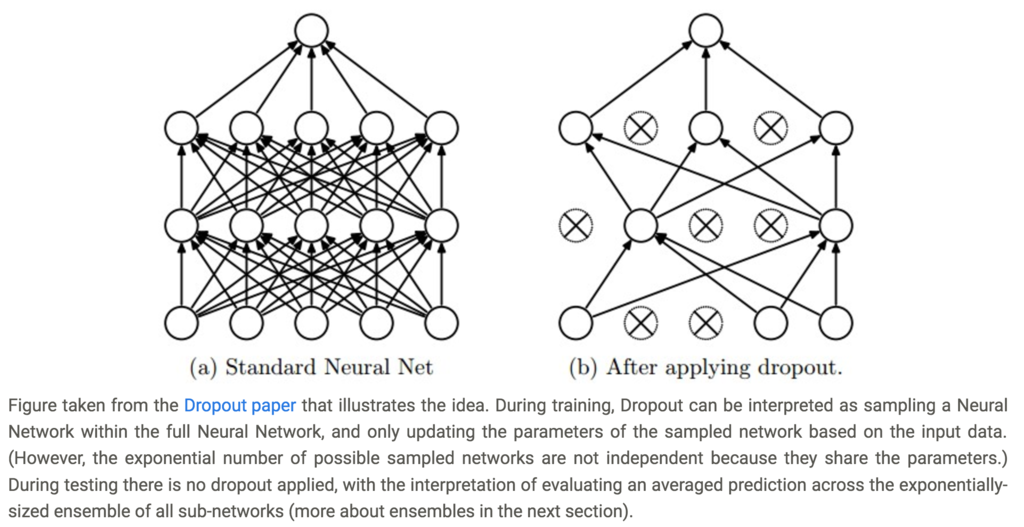 (CS231nより引用)
(CS231nより引用)
- dropoutのコード例
""" Vanilla Dropout: Not recommended implementation (see notes below) """ p = 0.5 # probability of keeping a unit active. higher = less dropout def train_step(X): """ X contains the data """ # forward pass for example 3-layer neural network H1 = np.maximum(0, np.dot(W1, X) + b1) U1 = np.random.rand(*H1.shape) < p # first dropout mask H1 *= U1 # drop! H2 = np.maximum(0, np.dot(W2, H1) + b2) U2 = np.random.rand(*H2.shape) < p # second dropout mask H2 *= U2 # drop! out = np.dot(W3, H2) + b3 # backward pass: compute gradients... (not shown) # perform parameter update... (not shown) def predict(X): # ensembled forward pass H1 = np.maximum(0, np.dot(W1, X) + b1) * p # NOTE: scale the activations H2 = np.maximum(0, np.dot(W2, H1) + b2) * p # NOTE: scale the activations out = np.dot(W3, H2) + b3
- inverted dropoutを使ったほうがコードがシンプルになる
- training時にのみdropoutし、test時のforward passはdropoutしない
""" Inverted Dropout: Recommended implementation example. We drop and scale at train time and don't do anything at test time. """ p = 0.5 # probability of keeping a unit active. higher = less dropout def train_step(X): # forward pass for example 3-layer neural network H1 = np.maximum(0, np.dot(W1, X) + b1) U1 = (np.random.rand(*H1.shape) < p) / p # first dropout mask. Notice /p! H1 *= U1 # drop! H2 = np.maximum(0, np.dot(W2, H1) + b2) U2 = (np.random.rand(*H2.shape) < p) / p # second dropout mask. Notice /p! H2 *= U2 # drop! out = np.dot(W3, H2) + b3 # backward pass: compute gradients... (not shown) # perform parameter update... (not shown) def predict(X): # ensembled forward pass H1 = np.maximum(0, np.dot(W1, X) + b1) # no scaling necessary H2 = np.maximum(0, np.dot(W2, H1) + b2) out = np.dot(W3, H2) + b3
- biasの正規化は一般的でない
- 実用上は、L2正規化とdropoutをともに利用するのが一般的
Loss functions
問題が色々ある
分類クラスが多すぎる問題
- 英単語やImageNetの場合などはラベルが多い
- Hierachical Softmaxを使うべき
- ラベルを木構造化
- これまでは一つの答えを選ぶ問題のみ扱ってきた。でもbinary vectorを算出する問題だったら?
Summary
- オススメの前処理
- データを中央に寄せ、その平均を0にする
- 特徴に沿って、データを[-1, 1]の範囲に正規化
- 重みを、標準偏差
をとるgaussian distributionとなるよう初期化
- numpyでは
python w = np.random.randn(n) * sqrt(2.0/n)
- numpyでは
- L2正規化とdropoutを使う
- batch normalizationを使う
理解できなかった内容
Sparse initialization. Another way to address the uncalibrated variances problem is to set all weight matrices to zero, but to break symmetry every neuron is randomly connected (with weights sampled from a small gaussian as above) to a fixed number of neurons below it. A typical number of neurons to connect to may be as small as 10.